Che cos’è la gene editing? Può essere una possibile terapia per le malattie istiocitarie come l’HLH?
Ce ne parla la Dott.ssa Fortunati in questo interessante articolo.
La terapia genica nello studio delle immunodeficienze primarie
Le immunodeficienze primarie sono oggetto da decenni di studi di terapia genica e il successo clinico in alcune di queste malattie rappresenta il culmine della sinergia tra clinici, ricercatori ed industrie farmaceutiche che ha migliorato la comprensione delle singole patologie e incrementato la conoscenza della biologia delle cellule staminali, della virologia e della genetica molecolare.
L’approccio classico di terapia genica consiste nell’utilizzo di virus patologici inattivati il cui genoma viene in gran parte sostituito dalla sequenza genica del gene che si vuole correggere.
Uno di questi, il gammaretrovirus (γRV), è stato utilizzato per correggere la mancanza di un enzima l’adenosina deaminasi (AD) che provoca una immunodeficienza combinata grave (SCID).
Le prime prove di terapiagenica
L’ADA-SCID in quanto malattia monogenica, è stata la prima ad essere oggetto di terapia genica che ha prodotto, nel 2016, il primo farmaco di nuova generazione (Strimvelis™), una pietra miliare fondamentale verso l’obiettivo di fornire nuovi farmaci per le malattie orfane (1). Strimvelis™ consiste in una singola infusione di cellule staminali ematopoietiche del paziente geneticamente modificate con il gene AD corretto da utilizzare per il ripopolamento del midollo spinale del paziente.
Le prime prove di terapia genica iniziate negli anni ’90, come abbiamo visto, sono approdate al secondo decennio del nuovo millennio con nuovi vettori virali ingegnerizzati che forniscono un profilo di sicurezza ed efficienza di espressione migliorati, grazie all’uso di sequenze chimeriche e promotori interni dei geni target (1, 2).
La terapia genica nello studio dell’HLH
Anche la HLH è stata oggetto di terapia genica e la prima considerata è stata la FLH2.
Questa forma familiare è causata dalla deficienza della perforina, una proteina rilasciata nella sinapsi immunitaria che permette l’eliminazione di cellule infette (3).
Le mutazioni nel gene perforina (PRF1) rappresentano fino al 58% dei casi di FHL a seconda dell’etnia e rappresentano quindi un buon modello per studiare la fattibilità dei protocolli di terapia genica.
Il primo approccio clinico, è stato quello di correggere le cellule T (CD8+) di pazienti in vitro attraverso l’uso di vettori retrovirali e lentivirus e di reintrodurle in vivo in modelli animali per la HLH.
Pur dimostrando la correzione del difetto genico in cellule in vitro e in cavie, il livello di attecchimento delle cellule T richiesto per proteggere e correggere il difetto HLH nell’uomo non è mai stato stimato, determinando perciò la necessità di ulteriori studi clinici (4).
Terapia genica e FLH
Anche la FHL3 che rappresenta il 30-40% dei casi di FHL è stata target di studi di terapia genica (5,6 e blog marzo 2021).
Infatti essa è causata da mutazioni nel gene UNC13D, che codifica per la proteina Munc13-4 essenziale per l’attivazione ed esocitosi delle vescicole contenenti perforina; le cellule prive di proteina funzionale non possono degranulare adeguatamente dando origine al difetto di citotossicità che è responsabile del quadro clinico della HLH.
Nel 2020, nuovi vettori lentivirali sono stati utilizzati per stabilire la soglia chimerica minima necessaria per ripristinare un’adeguata degranulazione e per dimostrare che il vettore genico descritto può essere utilizzato per trasferire i geni corretti (trasdurre) le cellule T dei pazienti FHL3 in vivo.
Questo si traduce nella raccolta di cellule T autologhe iper-attivate, la modificazione genica extra corporea, la loro re-infusione nel paziente (5) e attecchimento.
Inoltre alcuni aspetti di sicurezza biologica dell’uso di vettori virali devono essere ulteriormente indagati e chiariti.
Il gene editing
Mentre la terapia genica muoveva i primi passi nella clinica, la scoperta negli anni ’90 delle nucleasi apre una modalità alternativa di ripristinare le copie alterate dei geni.
Si apre l’era del gene editing.
L’editing genetico si avvale di nucleasi altamente sito-specifiche, comprese le nucleasi Zinc Finger (ZFN), TALEN (TALEN) e brevi ripetizioni palindromiche raggruppate regolarmente interspaziate e associate a sistemi proteici CRISPR (CRISPR/Cas).
Le nucleasi sono enzimi che come forbici molecolari tagliano, in modo guidato, sequenze specifiche del DNA e utilizzano frammenti omologi (che hanno la stessa origine) per la riparazione del tratto alterato (riparazione HDR, vedi figura). Quindi l’editing rispetto alla terapia genica rimuove, dove serve pezzi di DNA, correggendone il difetto genetico ed evita l’inserzione nel genoma umane di sequenze di origine virale. Recentemente ricercatori inglesi coordinati dalla Prof.ssa Booth si sono cimentati nel gene editing della XLP1 (7), una malattia linfoproliferativa legata all’X causata da mutazioni o delezioni nel gene SH2D1A che codifica per una proteina adattatrice intracellulare SAP. Quest’ultima è essenziale per diversi processi immunitari chiave. Infatti la sua l’assenza altera il funzionamento del sistema immunitario e si manifesta clinicamente in modo variabile anche come HLH.
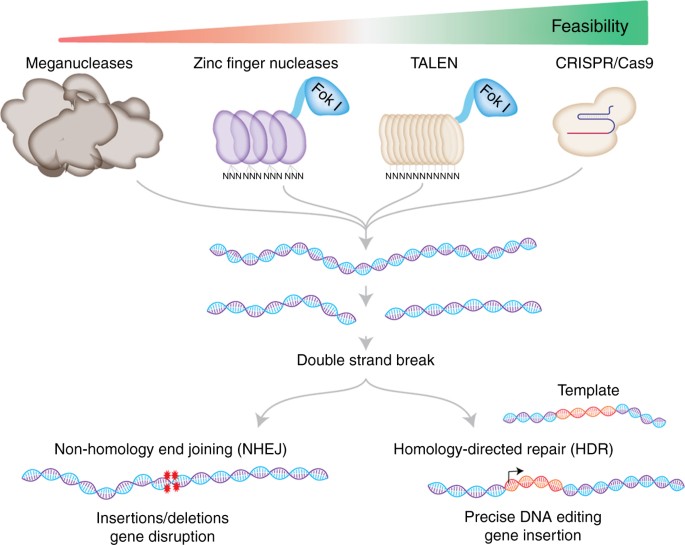
Tutti gli enzimi provocano dei tagli nella doppia elica che possono essere riparati con due modalità. La prima prevede la copiatura di un frammento omologo (HDR) che porta alla inserzione genica di una copia corretta del frammento.
La seconda una riparazione non omologa che porta all’inattivazione del gene.
Dall’articolo Mazhar Adli – review article: The CRISPR tool kit for genome editing and beyond DOI: 10.1038/s41467-018-04252-2
Il gene editing e la SAP
In un precedente lavoro il gruppo di scienziati aveva dimostrato che la terapia genica applicata in vitro a cellule T di pazienti XLP1 può correggere le anomalie di un topo modello carente di SAP (8) dimostrando la fattibilità della terapia genica per questa malattia. In questo nuovo articolo di recentissima pubblicazione, i ricercatori confrontano tre piattaforme di nucleasi attualmente disponibili (ZFN, TALEN, CRISPR/Cas) per correggere, attraverso il gene editing, la funzione delle cellule T del paziente.
Mediante l’utilizzo di una strategia che inserisce la copia corretta del gene SH2D1A nell’esone 1 in modo da preservare tutti gli elementi regolativi, e la trasduzione di tutti i componenti dell’editing attraverso un vettore virale (AAV6), i ricercatori hanno dimostrato in vitro, in cellule di 5 pazienti, la restaurazione dei livelli di espressione di SAP e conseguentemente delle capacità immunologiche delle cellule in almeno il 45 % dei linfociti T.
Inoltre le 3 piattaforme di editing non sono risultate dissimili tra loro per quanto riguarda l’espressione di SAP né nel numero di sequenze SAP extra geniche.
Sebbene, il numero di cellule T corrette necessarie per il beneficio clinico possa essere determinato solo nella sperimentazione clinica, possiamo augurarci che tale risultato possa essere presto utilizzato per esplorare la fattibilità del gene editing in altre malattie rare per esempio la HLH.
AILE supporta con impegno e dedizione il lavoro di medici e ricercatori per il raggiungimento di questi obiettivi.
Bibliografia
- Aiuti A, Roncarolo MG, Naldini L. Gene therapy for ADA-SCID, the first marketing approval of an ex vivo gene therapy in Europe: paving the road for the next generation of advanced therapy medicinal products. EMBO Mol Med. 2017 Jun;9(6):737-740.
- Houghton BC, Booth C. Gene Therapy for Primary Immunodeficiency. Hemasphere. 2020 Dec 29;5(1):e509.
- Ghosh S, Carmo M, Calero-Garcia M, Ricciardelli I, Bustamante Ogando JC, Blundell MP, Schambach A, Ashton-Rickardt PG, Booth C, Ehl S, Lehmberg K, Thrasher AJ, Gaspar HB. T-cell gene therapy for Perforin deficiency corrects cytotoxicity defects and prevents hemophagocytic lymphohistiocytosis manifestations. J Allergy Clin Immunol. 2018 Sep;142(3):904-913.e3.
- Panchal N, Ghosh S, Booth C. T cell gene therapy to treat immunodeficiency. Br J Haematol. 2021 Feb;192(3):433-443.
- Takushi SE, Paik NY, Fedanov A, Prince C, Doering CB, Spencer HT,Chandrakasan S. Lentiviral Gene Therapy for Familial HemophagocyticLymphohistiocytosis Type 3, Caused by UNC13D Genetic Defects. Hum Gene Ther. 2020 Jun;31(11-12):626-638
- Dettmer V, Bloom K, Gross M, Weissert K, Aichele P, Ehl S, Cathomen T. Retroviral UNC13D Gene Transfer Restores Cytotoxic Activity of T Cells Derived from Familial Hemophagocytic Lymphohistiocytosis Type 3 Patients In Vitro. Hum Gene Ther. 2019 Aug;30(8):975-984.
- Houghton BC, Panchal N, Haas SA, Chmielewski KO, Hildenbeutel M, Whittaker T, Mussolino C, Cathomen T, Thrasher AJ, Booth C. Genome Editing With TALEN, CRISPR-Cas9 and CRISPR-Cas12a in Combination With AAV6 Homology Donor Restores T Cell Function for XLP. Front Genome Ed. 2022 May 23;4:828489.
- Panchal, N., Houghton, B., Diez, B., Ghosh, S., Ricciardelli, I., Thrasher, A. J., et al. Transfer of Gene Corrected T Cells Corrects Humoral and Cytotoxic Defects in X-Linked Lymphoproliferative Disease (XLP1). J. Allergy Clin. Immun. 2018: 142, 235–245.
