La predisposizione genetica
La predisposizione o suscettibilità genetica, indica una serie di caratteristiche genetiche che rendono possibile il manifestarsi di una malattia. L’analisi delle informazioni sulla predisposizione genetica alla HLH contribuisce a formare modelli di ereditarietà utilizzati per spiegare la grande variabilità fenotipica con cui si presenta questa malattia.
Sono trascorsi quasi 23 anni dalla scoperta del primo gene associato alla HLH ed è giunto il tempo di riconsiderare in maniera critica i modelli di eredità proposti alla luce di nuove evidenze scientifiche.
È quello che la Dr.ssa Lauren Meyer dell’Università della California e la Dr.ssa Kim Nichols dell’ospedale Pediatrico St. Jude di Memphis hanno dettagliato in una recente pubblicazione ricapitolando i dati bibliografici degli ultimi anni (1).
Le due ricercatrici partendo dal modello Monogenico Classico, ne mettono in luce l’inadeguatezza, favoriscono quello più recente a due geni (Modello Digenico) e sostengono un Modello “Soglia” più adatto a spiegare le complesse interazioni tra predisposizione genetica e influenza ambientale nello sviluppo della HLH.
Modello Monogenico Classico (MMC)
È stato il primo, in ordine temporale che ha descritto l’HLH familiare (fHLH) come una malattia autosomica recessiva causata da due varianti germinali che determinano la perdita di funzione del gene alterato (LOF, abbr. dall’inglese Loss of Function). Storicamente è PRF1 il primo gene in cui mutazioni uguali (omozigosi) e mutazioni diverse tra loro (eterozigosi) nei due alleli (forme alternative dello stesso gene) sono state identificate nei pazienti HLH (2). Da allora si sono individuate varianti in altri geni quali UNC13D, STX11, STXBP2, LYST, AP3B1, e RAB27A. È subito parso evidente che tutti questi geni sono coinvolti nel corretto assemblaggio, esocitosi e funzione dei granuli citotossici presenti nei linfociti citotossici CD8+ e nelle cellule Natural killer.
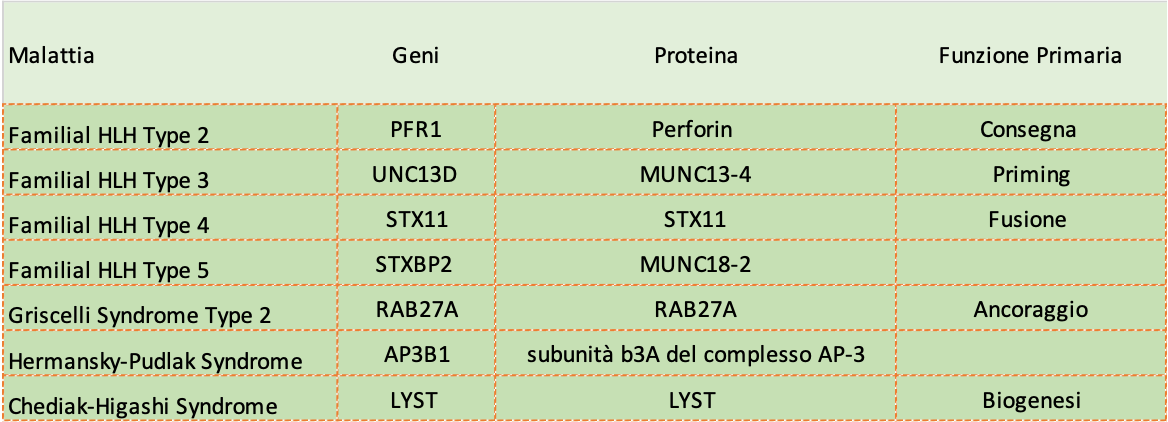
Come già illustrato in blog precedenti, i granuli citotossici contengono enzimi che entrano nella cellula bersaglio infetta, inducendone la morte. Il successo di questo meccanismo di difesa richiede l’attività coordinata delle numerose proteine codificate dai sopracitati geni. I diversi tipi di HLH, i geni e le proteine coinvolte nella via metaboliche che portano alle disfunzioni del sistema immunitario sono illustrate nella tabella. Varianti geniche in uno qualsiasi di questi geni causano anomalie immunitarie che hanno come conseguenza il manifestarsi della HLH e delle relative sindromi.
Il MMC interpreta bene i meccanismi di predisposizione della fHLH familiare, dove il 70% dei casi si verificano entro l’anno di vita o durante la prima infanzia, ma è incapace di spiegare quei casi clinici mancanti di una chiara causa genetica in cui è invece evidente l’alterazione del sistema immunitario. Un esempio è la HLH secondaria (sHLH) che si presenta più comunemente nella tarda infanzia o nell’età adulta e sviluppa un’iper-infiammazione in seguito all’esposizione a stimolo immunogenico forte come un’infezione o un tumore maligno.
Modello Di-Genico (MdiG)
Per superare la dicotomia tra HLH familiare e secondaria e le difficoltà crescenti del MMC nell’includere pazienti HLH con differenti meccanismi genetici di eredità, si è proposto un Modello Digenico.
Questo considera le varianti geniche germinali in un singolo gene non più sufficienti ma ritiene che, alla manifestazione della HLH possano concorrere varianti singole LOF in due geni distinti (3,4). Come esemplificazione consideriamo le varianti LOF classiche nel solo gene LYST o nel gene AP-3 che provocano rispettivamente le sindromi di Chediak-Higashi e di Hermansky-Pudlak.
Oltre a condividere alcune caratteristiche cliniche come l’albinismo, la propensione alle emorragie che riflettono dei meccanismi patofisiologici comuni, queste sindromi sviluppano segni e sintomi di HLH in risposta a infezioni.
Se consideriamo la funzione dei geni LYST e AP-3, noteremo che entrambi sono necessari alla biogenesi e all’assemblaggio dei granuli: LYST ne regola la dimensione, mentre il complesso AP-3 permette la corretta localizzazione in questi granuli delle proteine lisosomiali chiave che sono a loro volta necessari per la loro corretta funzione.
Ora consideriamo varianti singole LOF, cioè quelle che di per sé non sono sufficienti alla manifestazione della malattia, presenti però in entrambi i geni. Alterazioni geniche singole in LYS in aggiunta a quelle in AP-3 producono granuli citotossici inadeguati in dimensione a cui si somma l’incapacità di inserire correttamente le proteine lisosomiali impedendo l’appropriata degranulazione e funzione citotossica.
Il risultato complessivo è il fallimento della rimozione e la persistenza dell’insulto immunitario che scatena l’iperinfiammazione.
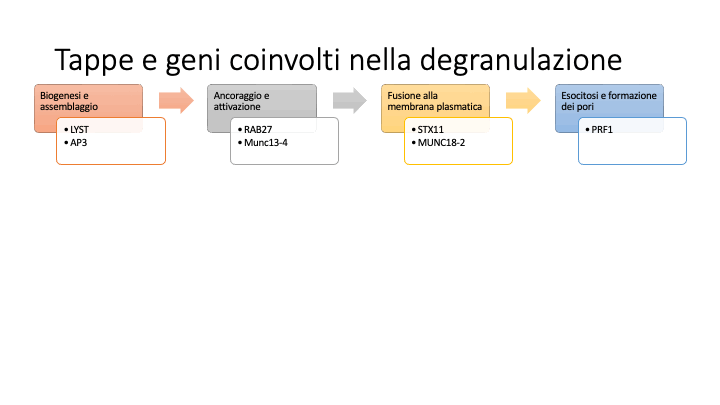
Altri esempi di MdiG si possono identificare in altre tappe della degranulazione come illustrato in figura (1) e riportate nelle voci bibliografia 5, 6, 7.
Modello Soglia (MS)
Un nuovo modello che risponda adeguatamente alle complesse interazioni tra la predisposizione genetica e l’influenza dell’ambiente, è quello chiamato Modello Soglia.
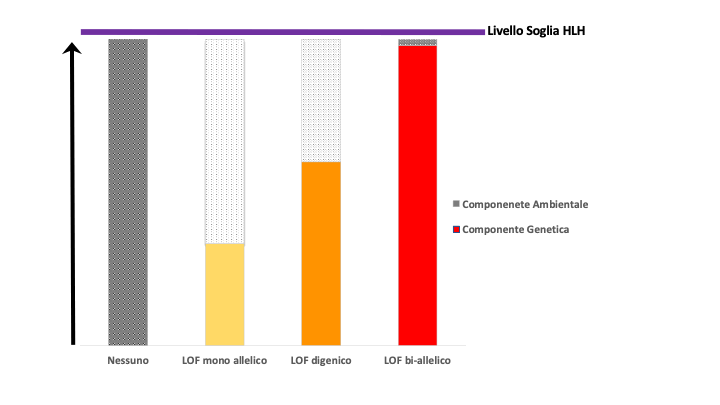
Esso prevede che l’HLH si manifesti quando si raggiunge un livello soglia che è dato dalla somma tra la predisposizione genetica e fattori ambientali.
Esiste cioè una relazione inversa tra il grado di disfunzione della via che conduce alla degranulazione lisosomiale, determinata dalle varianti germinali, e l’insulto immunogenico che scatena la HLH (Fig. 2 in basso): alterazioni ereditate gravi nelle vie metaboliche della degranulazione (LOF bi-allelico) sono sufficienti a determinare il fenotipo HLH che viene innescato da stimoli immunogenici lievi o assenti (barra rossa, a destra della Fig. 2 in basso).
Al contrario, alterazioni geniche più subdole nelle vie della degranulazione (LOF mono allelico o assente) richiedono uno stimolo immunogenico forte a cui segue un’intensa risposta immunogenica che induce l’iperinfiammazione (barra grigia a sinistra della Fig.2). Questo modello è stato inizialmente proposto da Jessen et al., (8) dopo che, studi condotti su topi in cui i geni della via lisosomiale erano stati silenziati, si era osservato uno spettro di gravità della malattia correlato all’entità del difetto di citotossicità.
Sulla base di questa analisi, il gruppo di ricercatori hanno proposto una gerarchia dei geni della via di degranulazione, con mutazioni in Ap3b1 che conferiscono il fenotipo più lieve seguito da Lyst, Stx11, Rab27a e infine Prf1.
Questa stessa gerarchia è stata successivamente confermata dall’analisi di una coorte di pazienti con LOF completa in questi stessi geni (9).
Conclusioni
Lo studio riportato della Dr.ssa Meyer e Nichols su 44 pazienti HLH e incluso nella pubblicazione (1), mette in evidenza la necessità di comprendere meglio l’impatto di combinazioni di varianti sulla predisposizione all’HLH attraverso ricerche più complete in cui il sequenziamento esomico e l’analisi funzionali e biochimiche delle varianti associate, venga correlato a fattori ambientali scatenanti. Nonostante gli sforzi della comunità scientifica siamo ben lungi dall’aver compreso completamente la predisposizione genetica nella HLH e quale sia il reale contributo dell’ambiente nel determinare il manifestarsi e la gravità di questa sindrome immunitaria.
Bibliografia
- Steen EA, Hermiston ML, Nichols KE, Meyer LK. Digenic Inheritance: Evidence and Gaps in Hemophagocytic Lymphohistiocytosis. Front Immunol. 2021 Nov 17; 12:777851
- Stepp SE, Dufourcq-Lagelouse R, Le Deist F, Bhawan S, Certain S, Mathew PA, Henter JI, Bennett M, Fischer A, de Saint Basile G, Kumar V. Perforin gene defects in familial hemophagocytic lymphohistiocytosis. Science. 1999 Dec 3;286(5446):1957-9.
- Schäffer AA. Digenic inheritance in medical genetics. J Med Genet. 2013 Oct;50(10):641-52.
- Ameratunga R, Woon ST, Bryant VL, Steele R, Slade C, Leung EY, Lehnert K. Clinical Implications of Digenic Inheritance and Epistasis in Primary Immunodeficiency Disorders. Front Immunol. 2018 Jan 26;8:1965.
- Zhang M, Bracaglia C, Prencipe G, Bemrich-Stolz CJ, Beukelman T, Dimmitt RA, Chatham WW, Zhang K, Li H, Walter MR, De Benedetti F, Grom AA, Cron RQ. A Heterozygous RAB27A Mutation Associated with Delayed Cytolytic Granule Polarization and Hemophagocytic Lymphohistiocytosis. J Immunol. 2016 Mar 15;196(6):2492-503.
- Tang BL. SNAREs and developmental disorders. J Cell Physiol. 2021 Apr;236(4):2482-2504.
- Sheng L, Zhang W, Gu J, Shen K, Luo H, Yang Y. Novel mutations of STXBP2 and LYST associated with adult haemophagocytic lymphohistiocytosis with Epstein-Barr virus infection: a case report. BMC Med Genet. 2019 Feb 19;20(1):34.
- Jessen B, Kögl T, Sepulveda FE, de Saint Basile G, Aichele P, Ehl S. Graded Defects in Cytotoxicity Determine Severity of Hemophagocytic Lymphohistiocytosis in Humans and Mice. Front Immunol (2013) 4:448.
