Come si effettua l’identificazione delle varianti germinali
Una delle sfide nella HLH familiare (fHLH) è sicuramente quella di giungere ad una rapida ed accurata diagnosi. Nei pazienti affetti da fHLH, le cellule T (CD8 T) e le Natural Killer (NK) diventano attive in conseguenza di uno stimolo immunitario di poca o nessuna rilevanza. Queste cellule però sono incapaci di eliminare il target a causa di mutazioni (varianti) nei geni deputati al controllo di questa funzione, causando così l’iper infiammazione.
L’identificazione di almeno una variante germinale che produca la perdita di funzionalità (loss of function, LOH) delle cellule CD8 T e delle NK, diventa fondamentale per un esito terapeutico positivo dei pazienti. È quindi cruciale avere dei test funzionali che misurino l’impatto delle varianti sulla attività delle cellule immunitarie.
Attualmente, uno di questi test (1) misura la capacità delle cellule immunitarie di traslocare sulla superficie cellulare una proteina chiamata CD107a. Se ciò non avviene i linfociti non sono in grado di degranulare e dunque di compiere la loro funzione di eliminazione del target. Questo avviene in presenza di varianti germinali in alcuni geni che sono noti per causare la fHLH. Per cui sfruttando questo test si può desumere la presenza di mutazioni nei geni UNC13D, STX11, e STXBP2, ma non in PFR1.
Un altro test funzionale complementare è quello che usa le cellule NK del paziente per uccidere delle cellule in vitro: purtroppo l’uso di questo test è limitato dalla ridotta specificità e sensibilità (2) poiché l’iper infiammazione può reprimere l’attività citotossica delle cellule NK.
Un nuovo approccio diagnostico che supera la complessità genetica e i problemi legati ai test funzionali è stata recentemente pubblicata sulla rivista Blood da un gruppo australiano (3). La nuova procedura ha come scopo quello di identificare soprattutto varianti di “significato incerto” le cosiddette VUS (Variant of Uncertain Significance) nei geni fHLH e capirne l’effetto.
Un nuovo approccio
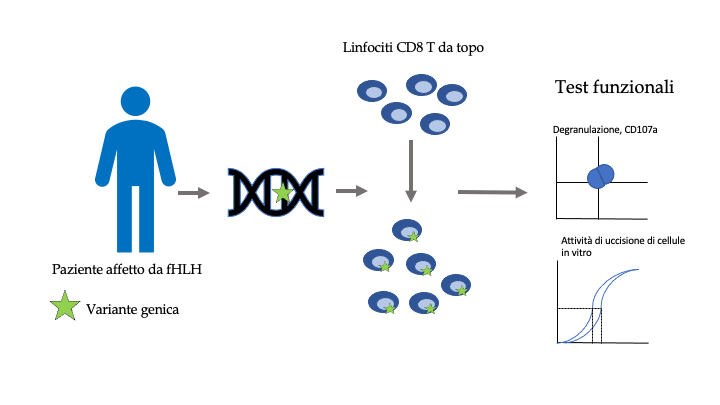
Inizialmente si isolano linfociti CD8 T da un modello animale che in questo caso è il topo. Successivamente, utilizzando la tecnica di genome editing detta CRISPR/CAS, si inattiva nelle CD8 T, il gene murino corrispondente a quello umano di interesse, per esempio il gene che porta la variante genica identificata nell’uomo. La conseguenza di questa inattivazione è la compromissione della capacità delle cellule di topo di traslocare la proteina CD107a sulla superficie delle cellule T e quindi di indurre l’eliminazione del target immunitario. Nel frattempo si genera in laboratorio un vettore di espressione, una sequenza genica umana di DNA (cDNA) che contiene le varianti identificate nel paziente e che è complementare a quella inattivata nel genoma delle cellule di topo. Il cDNA umano viene successivamente inserito nelle cellule CD8 T murine e dopo aver confermato la capacità di produrre una proteina, si procede alla valutazione della traslocazione della CD107a e della loro capacità citotossica. Se le varianti umane nei geni fHLH sono patogeniche non ci sarà né traslocazione né capacità citossiche delle cellule CD8 T di topo. Viceversa se le varianti del paziente non impediscono una funzione residua o completa dei geni fHLH, si registrerà una attività in entrambi i parametri. In questo caso si può desumere che le varianti non sono patogeniche.
Conclusioni
Questo nuovo test rappresenta sicuramente un avanzamento nell’ identificazione e nel riconoscimento del significato biologico delle VUS, e nella accelerazione della procedura diagnostica. Ci sono però degli aspetti del test funzionale che pongono delle domande che necessitano di ulteriori approfondimenti. Un aspetto è legato alla laboriosità del test e all’utilizzo di competenze tecnologiche che non sono disponibili in tutti i laboratori, rendendo difficile perciò l’applicazione della metodica in un set di tipo clinico. Altra considerazione è che i risultati vanno attentamente interpretati alla luce della possibilità che l’inattivazione dei geni murini sia incompleta, e di una espressione variabile della proteina introdotta. Infine il test permette di ricercare le conseguenze funzionali di una singola variante alla volta, mentre sappiamo che molti pazienti fHLH sono portatori di varianti in eterozigosi, che colpiscono uno o due geni. A questo dobbiamo aggiungere che alcuni risultati riportati nell’articolo di Noori et al., sottolineano delle differenze biologiche tra l’uomo e il topo che impongono approfondimenti di studio per poter traslare nell’uomo i risultati ottenuti con le cellule CD8 T murine. Tuttavia questi nuovi approcci tecnologici come l’editing sembrano molto promettenti per comprendere meglio le cause genetiche della fHLH e l’identificazione di mutazioni ancora non conosciute.
Bibliografia
- Rubin TS, Zhang K, Gifford C, et al. Perforin and CD107a testing is superior to NK cell function testing for screening patients for genetic HLH. Blood. 2017;129(22):2993-2999.
- Jang HS, Flinsenberg TWH, Lacaze P, et al. Recovery of natural killer cell cytotoxicity in a p.A91V perforin homozygous patient following severe haemophagocytic lymphohistiocytosis. Br J Haematol. 2020; 190(3):458-461.
- Noori T, Rudd-Schmidt JA, Kane A, Frith K, Gray PE, Hu H, Hsu D, Chung CWT, Hodel AW, Trapani JA, Voskoboinik I. A cell-based functional assay that accurately links genotype to phenotype in familial HLH. Blood. 2023 May 11;141(19):2330-2342.
